Nuevos roles y retos del farmacéutico de hospital. New roles and challenges of the hospital pharmacist
Más datosel objetivo de este artículo es realizar una revisión narrativa de cómo se está aplicando la farmacogenética y la farmacogenómica en la clínica, especialmente en España.
Métodose han revisado las publicaciones y sitios web de mayor interés.
Resultadosse discuten los farmacogenes y variantes en uso en varios centros hospitalarios, las metodologías empleadas y el proceso de implementación.
The aim of this article is to perform a narrative review of how pharmacogenetics and pharmacogenomics is being applied in the clinic, especially in Spain.
MethodPublications and websites of major interest have been reviewed.
ResultsPharmacogenes and variants used in several hospitals, available methodologies, and the implementation process are discussed.
La identificación y aplicación de la estrategia terapéutica más eficaz para cada paciente parece que se puede alcanzar gracias al avance científico y tecnológico, y es el objetivo de la mayoría de los sistemas de salud1,2. Surge así el concepto de medicina de precisión como la identificación y aplicación de la estrategia terapéutica, diagnóstica y preventiva más eficaz para cada paciente o subgrupo de población, teniendo en cuenta la información genética y la influencia del medio ambiente3. En la parte que toca a la farmacia, este concepto supone la evaluación de la genómica y exposición ambiental, el estilo de vida y el análisis de otras características únicas del paciente o de la enfermedad para orientar la selección y dosificación de los fármacos4,5. En ambos casos la protagonista es la genética o la genómica, entendida como la suma de la genética y otros elementos presentes en el genoma fuera de los genes. Así surgen la farmacogenética y la farmacogenómica. En este artículo usaremos la abreviatura PGx para referirnos a ellas de forma indistinta, ya que los objetivos básicamente son los mismos.
La aplicación de la PGx en la sociedad actual, donde el consumo de fármacos es muy elevado6, se supone debería tener un impacto enorme. Por una parte, puede evitar los efectos adversos que requieren atención y en muchos casos hospitalización, y por otra, seleccionar el tratamiento, eliminando los que son ineficaces. Además, la PGx puede usarse para anticipar el tratamiento, genotipando a la población7. De hecho, no es raro que cualquier persona tenga alguna variante genética relacionada con la PGx y tampoco que esa persona se vea expuesta a varios medicamentos a lo largo de su vida y, a veces, a la misma vez. En este último sentido, la PGx podría conducir a prevenir los efectos adversos relacionados con la polifarmacoterapia.
A tenor de todo lo anterior, la implementación de la PGx debería ser generalizada; sin embargo, no parece serlo. Por tanto, nos pareció de interés conocer la aplicación de la PGx en la práctica clínica habitual en España (genes, variantes y metodología) y los elementos que intervienen en su implementación.
MétodosLa revisión se ha realizado a varios niveles: 1) bibliografía, revisión no sistemática de los últimos 10 años en PubMed, WOS. Como términos de búsqueda se usaron los siguientes: «Farmacogenética», «Farmacogenómica», «Medicina de Precisión», «Medicina Personalizada», «Farmacoterapia de Precisión», «Farmacoterapia Personalizada» y sus correspondientes en inglés: «Pharmacogenetics», «Pharmacogenomics», «Precision Medicine», «Personalized Medicine», «Precision Pharmacotherapy», «Personalized Pharamacotherapy». 2) Bases de datos: Pharmacogenomics Knowledge Base (PharmGKB)8, Pharmacogene Variation (PharmVar)9 y ClinVar10. PharmGKB contiene los farmacogenes que han sido aprobados por la US Food and Drug Administration (FDA)11,12, European Medicines Agency (EMA)13, Swiss Agency of Therapeutic Products (Swissmedic)14, Pharmaceuticals and Medical Devices Agency, Japan (PMDA)15 y Health Canada (Santé Canada) (HCSC)16. Pharmvar contiene fundamentalmente variantes del CYP450. Por último, ClinVar contiene farmacogenes y otros biomarcadores, siendo la referencia para la relación genotipo-fenotipo en los humanos. 3) Guías clínicas: Clinical Pharmacogenetics Implementation Consortium (CPIC)17 (cpicpgx.org) por la parte americana y Dutch Pharmacogenetics Working Group (DPWG) por la parte europea18–22. 4) Instituciones y organizaciones españolas: Consejo General de Colegios de Farmacéuticos (https://www.farmaceuticos.com/), Sociedad Española de Farmacia Hospitalaria (https://www.sefh.es/), Sociedad Española de Farmacogenética y Farmacogenómica (https://seff.es/), Sociedad Española de Farmacia Clínica, Familiar y Comunitaria (https://www.sefac.org/).
ResultadosGenes, variantes y metodologíaLos farmacogenes y metodologías en uso son muy similares en todos los países analizados. En la tabla 1, se indican los farmacogenes más utilizados. Se indican 2 ejemplos españoles elegidos por la disponibilidad de información detallada. El 16% de los farmacogenes que se analizan son proteínas de transporte; el 39% proteínas del metabolismo y el 45% dianas terapéuticas. Así, los farmacogenes de biodisponibilidad (transporte y metabolismo) suponen el 55% frente al 45% de dianas terapéuticas. Los tratamientos afectados muestran que las principales áreas terapéuticas son la antitumoral (23%), la neurológica (18%), los inmunosupresores (11%), los antirretrovirales (10%), los anticoagulantes (8%), los antihipercolesterolemiantes (8%), los antidiabéticos (8%) y las terapias biológicas (5%). Estos resultados concuerdan con los representados en la figura 1 relativa a los servicios en que se oferta la PGx.
Farmacogenes usados en proyectos e iniciativas en España y el extranjero
| CPIC+DPWG | Sanford chip | PriME-PGx | La Paz | rsID (variantes) | Familia de proteínas | Función | Tratamientos afectados |
|---|---|---|---|---|---|---|---|
| ABCB1 | ABCB1 | rs2032582; rs1045642; rs3213619; rs1128503; | ATP Binding Cassette | T | Inmunosupresores y antiagregantes plaquetarios | ||
| ABCC2 | rs717620; rs56296335; rs3740066; rs56199535; rs56220353; | ATP Binding Cassette | T | Antiretrovirales | |||
| ABCG2 | ABCG2 | rs2231142; rs2273697; rs72552713; | ATP Binding Cassette | T | Estatinas, metotrexato, imatinib (inhibidor tirosina-cinasa) | ||
| APOE | rs7412 | Apolipoproteínas (chaperonas) | D | Anticoagulantes, pravastatina | |||
| CACNA1S | rs1800559; rs772226819; | Receptor dihidropiridina | D | Estatinas | |||
| COMT | COMT | rs4680 | Catecol-O-metiltransferasa | M | Catecolaminas y derivados | ||
| CFTR | CFTR | rs75527207; rs113993960; rs199826652; rs267606723; rs193922525; rs80282562; rs121909013; rs74503330; rs121909041; rs121908755; rs121909005; rs121908757; | Regulador de la conductabilidad transmembrana de la fibrosis quística | D | Ivacaftor | ||
| CYP1A2 | rs2069514; rs762551; rs2470890; | Citocromo P450 | M | Fenacetina, cafeína,clozapina, tacrina, propranolol | |||
| CYP2A6 | rs28399433 | Citocromo P450 | M | Letrozol, tegafur, cumarinas, valproico, metoxifluorano, disulfiram, halotano, fadrozol | |||
| CYP2B6 | CYP2B6 | rs374527407; rs3211371; rs4803419; rs2279343; rs34223104; rs28399499 | Citocromo P450 | M | Artemisinina, bupropion, ciclofosfamida, efavirenz, ketamina, metadona | ||
| CYP2C19 | CYP2C19 | CYP2C19 | CYP2C19 | rs4244285; rs4986893; rs12248560; rs28399504; rs56337013; rs72552267; rs72558186; rs41291556; rs267606723; rs193922525; rs80282562; rs121909013; rs74503330; rs121909041; rs121908755; rs121909005; rs121908757; | Citocromo P450 | M | Antifúngicos y antiagregantes plaquetarios |
| CYP2C8 | CYP2C8 | Rs11572080; rs10509681; rs1058930; rs11572103; | Citocromo P450 | M | Paclitaxel, psicotropos, antidiabeticos orales | ||
| CYP2C9 | CYP2C9 | CYP2C9 | CYP2C9 | rs1799853; rs1057910; | Citocromo P450 | M | Psicotropos, anticonvulsivantes |
| CYP2D6 | CYP2D6 | CYP2D6 | CYP2D6 | rs1080985; rs28371725; rs35742686; rs3892097; rs5030655; rs5030865; rs5030867; rs5030656; rs1065852; rs1058164; rs1135840; rs16947; rs28371706; rs61736512; rs769258 | Citocromo P450 | M | Psicotropos, opioides antináuseas (ondansetron) |
| CYP3A4 | CYP3A4 | rs55785340; rs4646438; | Citocromo P450 | M | Inmunosupresores | ||
| CYP3A5 | CYP3A5 | CYP3A5 | CYP3A5 | rs776746; rs55965422; rs10264272; rs41303343; rs41279854 | Citocromo P450 | M | Tacrolimus |
| CYP4F2 | CYP4F2 | CYP4F2 | CYP4F2 | rs2108622; | Citocromo P450 | M | Anticoagulantes |
| DPYD | DPYD | DPYD | DPYD | rs3918290; rs55886062; rs67376798; rs1801159; rs1801265; | Dihidropirimidina deshidrogenasa | M | Fluoropirimidinas (5FU) |
| ERCC1 | ERCC1 | rs11615; rs3212986 | Endonucleasa de reparación por escisión | D | Cisplatino | ||
| EPHX1 | EPHX1 | rs1051740 | Endonucleasa de reparación por escisión | M | Cisplatino | ||
| FCGR2A | rs1801274; | Epóxido hidrolasa | D | Inmunoterapia | |||
| G6PD | rs782669677 | Glucosa 6 fosfato deshidrogenasa | M | Primaquina | |||
| HLA-A3101 | rs1061235 | Complejo mayor histocompatibilidad | D | Carbamacepina, oxcarbazepina, lamotrigina, fenitoína | |||
| HLA-B | rs144012689 | Complejo mayor histocompatibilidad | D | Carbamacepina, oxcarbazepina, lamotrigina, fenitoína | |||
| HCP5 | rs2395029 | Complejo mayor histocompatibilidad | D | Retrovirus | |||
| HTR2A | rs6311 | Receptor 5-hidroxitriptamina | D | Psicotropos. Serotonin 5-HT2A antagonistas | |||
| IFLN3 | IFLN3 | rs4803217 | Interferón | D | Hepatitis C | ||
| IFLN4 | rs469415590; | Interferón | D | Hepatitis C | |||
| IL10 | rs1800896 rs1800872; rs1800871 | Citoquina | D | Inmunoterapia antiinflamatoria | |||
| IL23R | rs7517847; rs10489629; rs11465804; rs1343151 | Receptor citoquina | D | Terapia biológica | |||
| IL28B | rs12979860 | Citoquina | D | Interferón. Ribaviria | |||
| KCNJ6 | rs2070995 | Canal de potasio | D | Metadona | |||
| MTHFR | MTHFR | rs1801133; rs4846051; rs1801131 | Metilén tetrahidrofolato reductasa | M | Metotrexato | ||
| NUDT15 | NUDT15 | rs116855232 | Nucleótido hidrolasa | M | Tiopurinas | ||
| OPRM1 | rs1799971 | Receptor opioide mu | D | Antidepresivos | |||
| POR | rs1057868; rs2868177 | Citocromo P450 | M | Immunosupresores | |||
| RARG | rs2229774 | Receptor ácido retinoico gamma | D | Doxorubicina | |||
| RYR1 | rs118192172 | Receptor rianodina | D | Estatinas | |||
| SLC15A2 | rs2293616; rs2257212; rs1143671; rs1143672 | Transportador H+/péptidos | T | Metformina | |||
| SLC22A1 | SLC22A1 | rs72552763; rs55918055; rs36103319; rs34059508; rs628031; rs4646277; rs2282143; rs4646278; rs12208357 | Transportador de cationes orgánicos | T | Tramadol, metformina | ||
| SLC22A2 | rs316019; rs8177516; rs8177517; rs8177507; rs8177504 | Transportador de cationes orgánicos | T | Fampridina, metformina | |||
| SLC22A6 | rs11568626 | Transportador de cationes orgánicos | T | Aciclovir, zidovudina, didanosina, zalcitabina, lamivudina, estavudina, trifluridina, cidofovir, adefovir, tenofovir | |||
| SLC28A3 | rs7853758 | Transportador Nucleósidos | T | Tiopurinas | |||
| SLCO1B1 | SLCO1B1 | SLCO1B1 | SLCO1B1 | rs4149056; rs2306283; rs56101265; rs72559745; rs56061388; rs55901008; rs59502379; rs56199088; rs55737008; rs4149015 | Transportador de aniones orgánicos | T | Estatinas, irinotecan, antidiabéticos orales, estrógenos |
| TBL1Y | rs768983 | Transducina beta | D | Terapias biológicas | |||
| TLR2 | rs4696480; rs11938228 | Receptores Toll | D | Inmunoterapias | |||
| TLR9 | rs352139 | Receptores Toll | D | Inmunoterapias | |||
| TNF | rs1800629 | Factor necrosis tumoral | D | Inmunoterapias | |||
| TP53 | rs1042522 | Supresor tumoral | D | Cisplatino | |||
| TPMT | TPMT | TPMT | TPMT | rs1800460; rs1800462; rs1142345; rs1800584 | Tiopurina metiltransferasa | M | Tioguaninas. 6-mercaptopurina y azatioprina |
| UGT1A1 | UGT1A1 | rs887829; rs4148323; rs34993780; rs35350960; rs55750087; rs4124874 | UDP-glucuronosil transferasa | M | Irinotecan. Acetaminofen, carvedilol, etopósido, lamotrigina. Simvastatina | ||
| UGT1A4 | rs2011425 | UDP-glucuronosil transferasa | M | Irinotecan. paracetamol, carvedilol, etopósido, lamotrigina. Simvastatina | |||
| UGT2B7 | rs7438135 | UDP-glucuronosil transferasa | M | Morfina, micofenolato | |||
| UGT2B15 | rs1902023 | UDP-glucuronosil transferasa | M | Benzodiacepinas | |||
| VKORC1 | VKORC1 | VKORC1 | VKORC1 | rs9934438 | Vitamina K epóxido reductasa | D | Anticoagulantes. Warfarina y acenocumarol |
| XPC | XPC | rs2228001 | Proteína de reparación del ADN | D | Cisplatino | ||
| XRCC1 | XRCC1 | rs25487 | Proteína de reparación del ADN | D | Cisplatino, inhibidores PPAR (olaparib, niraparib, rucaparib) |
CPIC: Clinical Pharmacogenetics Implementation Consortium17; D: Target; DPWG: Dutch Pharmacogenomic Working Group18–22; Sanford chip23: PriME-PGx, Hospital de la Princesa24; La Paz: Hospital de la Paz25; M: Metabolismo; rsID: reference SNP cluster identification, número identificación de la variante en dbSNP26; T: transporte.
Implementación de la PGx en áreas clínicas (España). Los valores representan el número de hospitales que ofertan el servicio de PGx. Los datos proceden de la SEFH27.
El número de variantes que se analizan para cada gen es relativamente pequeño, circunscribiéndose en la mayoría de los casos a uno. En la tabla se indican los números de referencia «rs», lo que permite acceder a la descripción de cada variante en la base de datos dbSNP26 (https://www.ncbi.nlm.nih.gov/snp).
La metodología mayoritaria es el análisis PCR y en menor medida los arrays (resultados no mostrados). En el caso de los arrays predominan los paneles de variantes formados ad hoc frente a los comerciales de composición fija, que son muy utilizados en otros países. La secuenciación NGS (Next Generation Sequencing) no se utiliza prácticamente, salvo en investigación. Se realizan 2 tipos: secuenciación WES (Wide Exome Sequencing), que analiza aproximadamente un 1-2% del genoma y es la más común, la WGS (Wide Genome Sequencing) que analiza el genoma completo. Un aspecto singular de la NGS es el número reads o fragmentos en que se divide el ADN, fragmentos que se secuencian simultáneamente (secuenciación masiva) y que luego hay que ordenar para obtener la secuencia de la muestra28. La NGS más usada es la que se realiza con reads largos de hasta 45.000 (LRS, Long-Reads Sequencing).
Implementación de la farmacogenética y la farmacogenómicaLa implementación en el ámbito hospitalario es amplia, mientras que en el ámbito comunitario es muy escasa. Esta situación es similar en otros países. En los hospitales se han creado unidades de farmacogenética que atienden las demandas de las áreas clínicas, especialmente la oncohematología (fig. 1)27. En las oficinas de farmacia, la aplicación es muy dispar e incipiente. No parece existir infraestructura como en los hospitales y las pruebas se encargan a laboratorios especializados.
El desarrollo de la PGx se está llevando a cabo por programas o proyectos específicos. En el extranjero, son los Estados Unidos los que disponen de mayor desarrollo, con programas como el Cleveland Clinic’s Personalized Medication Program, CLIPMERGE PGx initiative, eMERGE-PGx initiative, IGNITE, INGENIOUS, 1200 Patients Project and PREDICT29,30. En Europa, son los Países Bajos los más avanzados. De hecho, fueron los primeros en publicar guías clínicas (DPWG) y desarrollar sistemas donde se recopilan datos prospectivos sobre múltiples farmacogenes importantes y se incorporan a la historia clínica electrónica de los pacientes31. En España, cabe destacar los programas del Instituto de Salud Carlos III25, de la Sociedad Española de Farmacogenética y Farmacogenómica32, del Hospital de la Princesa24 y el proyecto MedeA33.
El desarrollo de la PGx en las distintas comunidades autónomas es dispar34 como se ilustra en la figura 2. Cuando se analizan los elementos clave de la implementación, se puede observar que el que más contribuye es la existencia de planes y estrategias gubernamentales, le siguen la colaboración público-privada y la formación. La existencia de masa crítica y de infraestructuras influyen mucho menos.
Medicina de precisión en España y elementos clave en su desarrollo. Los datos proceden del informe Roche34. 1) Cantabria, 2) Castilla La Mancha, 3) Islas Canarias , 4) Aragón, 5) La Rioja, 6) Principado de Asturias, 7) Islas Baleares, 8) Extremadura, 9) Comunidad Valencia, 10) Navarra, 11) Madrid, 12) Region de Murcia, 13) Castilla y León, 14) Galicia, 15) País Vasco, 16) Andalucía, y 17) Cataluña.
La PGx ha evolucionado desde las unidades específicas creadas en los hospitales para resolver problemas clínicos puntuales al estudio de paneles o secuenciación masiva de grupos poblacionales en laboratorios centralizados de ámbito regional y nacional7. En este sentido, es destacable la existencia del Collaborative Spanish Variability Server (CSVS)35, una base de datos que está recogiendo genomas y exomas de individuos y que permite conocer la prevalencia de variantes farmacogenómicas en la población española.
DiscusiónEl número de farmacogenes en uso es relativamente bajo cuando se compara con los genes considerados de utilidad o farmacogenes accionables (284 genes asociados con farmacocinética y 771 genes asociados a farmacodinamia)17–22. Igual ocurre con el número de variantes que se analizan. Esta discrepancia parece obedecer a 2 razones principales: la significación biológica y la metodología.
Por lo que respecta a la significación biológica, podemos distinguir 2 categorías de farmacogenes: los que afectan a la biodisponibilidad y los que afectan a las dianas terapéuticas. En el primer grupo la significación biológica es bastante clara, ya que estos farmacogenes tienen funciones concretas en la absorción, metabolismo, distribución y eliminación del fármaco, y así se suelen incluir en la mayoría de los protocolos. No obstante, las proteínas que participan en la biodisponibilidad tienen una especificidad de sustrato amplia, pudiendo utilizarse para varios fármacos, de ahí que se puedan utilizar los mismos paneles en diversas situaciones clínicas. Un aspecto digno de tener en cuenta es que muchas de las proteínas implicadas son inducibles, por lo que la ingestión del fármaco con el agente inducible (por ejemplo, alcohol u otro fármaco) puede generar efectos adversos como consecuencia de las variaciones en la cantidad de proteína. Tanto el carácter inducible como la especificidad de sustrato pueden tener efectos importantes en los enfermos polimedicados, un aspecto que debe tenerse en cuenta.
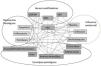
En el segundo caso las cosas no son tan claras, y así no es de extrañar que sean muchos los farmacogenes propuestos y pocos los elegidos. En principio, la diana terapéutica debe ser la proteína responsable del problema. Pero, en la mayoría de los casos, el problema es el resultado de un desequilibrio del sistema biológico, en el que intervienen más de una proteína, amén del efecto del ambiente sobre estas. En todos los casos nos enfrentamos a un problema multifactorial. A modo de ejemplo, en la figura 3 se muestra la red de factores e interacciones en el caso de la anemia falciforme. Como puede apreciarse, la alteración de la HbS, la principal responsable del problema, puede verse afectada por otras proteínas modificadoras, por la influencia ambiental y por la respuesta fisiológica de cada persona. En este caso deberían analizarse todos esos elementos, ya que originan distintos fenotipos clínicos susceptibles de tratamiento individualizado, pero en la práctica, cuando el efecto de alguno de estos factores es modesto, se suelen excluir. Otra razón que explica que no todas las dianas se utilicen es que en muchos casos se han identificado como asociaciones estadísticas y no funcionales, por lo que no está claro en qué forma afectan. Finalmente, el efecto del ambiente puede ser tan importante como los genes, ya que las proteínas pueden funcionar de forma diferente si varia, por ejemplo, el pH. En otras palabras, que los estudios genéticos no pueden considerarse definitivos a la hora de la toma de decisiones terapéuticas. Una forma de evaluar globalmente los sistemas biológicos es analizar el metaboloma, que representa el resultado de la acción de las proteínas y los efectos ambientales. En este sentido, se aprecia que cada vez es más frecuente incluir el análisis de metabolitos junto a los análisis genéticos.
Desde el punto de vista de la metodología se están asumiendo riesgos, ya que no siempre se analizan todas las posibles variantes. Sin embargo, es razonable usar arrays en los que se analiza un número limitado de variantes genómicas y dejar la secuenciación para casos especiales. Los casos especiales se refieren a variantes raras y variantes estructurales; los arrays no las detectan, ni tampoco permiten la asignación de las variantes a los alelos (haplotype phasing). Las variantes raras tienen un mayor impacto en la función y expresión de los genes y una mayor especificidad poblacional y desempeñan papeles importantes en la genética de enfermedades complejas36. Las variantes estructurales se refieren a deleciones, duplicaciones, inserciones, inversiones y translocaciones, así como reordenamientos complejos, todos ellos muy comunes en la PGx. En lo que se refiere al phasing, hay que recordar que, aunque se hable del genoma, en realidad cada persona tiene doble dotación cromosómica. Averiguar si las variantes están en uno u otro cromosoma puede hacer variar la respuesta a fármacos como el Alpelisib37.
La secuenciación más habitual es la de exomas completos (WES), la cual no analiza los intrones, en donde se han detectado variantes de interés en la PGx. Por otra parte, se está imponiendo la LRS (Long Read Sequencing) ya que a mayor tamaño de los fragmentos (reads), la secuencia establecida tiene menos errores. Estos errores provienen del hecho de que existen en el ADN muchas secuencias muy parecidas y por lo tanto, con reads cortos pueden haber varias alternativas. Por lo que respecta a la PGx son los de elección para el estudio de las variantes en genes complejos como el CYP2D6 o el HLA.
Implementación de la farmacogenética/farmacogenómicaLa implementación de la PGx se ha desarrollado en los hospitales, aunque es posible a todos los niveles. De hecho, buena parte de la PGx va dirigida a los fármacos de uso ambulatorio como los antidiabéticos, estatinas, etc. Si se analizan los elementos necesarios para establecer un servicio de PGx, que son según Cavallari et al.38: selección de los pacientes, procedimientos analíticos de genotipado, sistema informático, formación del personal y control de calidad, se comprende que la implementación sea en los hospitales, por tener más facilitades operacionales. Además, hay una razón histórica, y es que la mayoría de las iniciativas han surgido de proyectos de investigación, mayoritariamente en los hospitales. Es de suponer que la PGx se desarrolle más en el ámbito extrahospitalario y a ello puede contribuir la creciente tendencia del uso de paneles genéticos por Internet. En este sentido la FDA ha aprobado los DTC (direct-to-consumer), en que se evalúan marcadores de riesgo de cáncer (BRCA 1 y 2) y otras enfermedades (G6PDH). Como muchos de estos test se hacen sin prescripción, es evidente la necesidad de informar a la población, un aspecto en el que la farmacia comunitaria es clave.
Llama la atención que, siendo el avance en el conocimiento espectacular, la implementación de la PGx esté siendo tan lenta, tanto en España como en otros países39. Entre las causas que se han señalado para explicar este hecho se indican: un cierto escepticismo, la dificultad técnica, la falta de especialistas y el presupuesto31,40. Los críticos indican que con relación a los farmacogenes las cosas no están claras. Además, no siempre las pruebas de genotipado están estandarizadas ni las formas de registro. Más aún, para la implementación es importante el reconocimiento por las agencias de medicamentos, las cuales utilizan criterios diferentes. En nuestro caso habría que incluir al menos los medicamentos en los que la AEMPS/EMA recoge en sus fichas técnicas el análisis PGx. Por lo que respecta a la dificultad técnica, el problema recae más en la gestión de los resultados que en la analítica41. Así, la enorme cantidad de datos que deben analizarse para dar una solución requiere algoritmos matemáticos fiables, un aspecto que ha mejorado mucho con el desarrollo de la inteligencia artificial y una enorme capacidad computacional para dar con la solución a aplicar de forma rápida. Esto es un desafío ya que muchas de esas bases de datos no están estructuradas y no son interoperables, por lo que su integración es casi imposible. La parte analítica ha evolucionado desde las primeras técnicas casi artesanales a las actuales automatizadas fáciles de aplicar. Encontrar un laboratorio no es difícil. La SEFF está elaborando el mapa de laboratorios en España. En el extranjero la oferta es amplísima, pudiendo consultarse en la base de datos Genetic Testing Registry42. Los especialistas poco a poco van aumentando y por lo que respecta al costo, la tendencia es a la baja.
La implementación de la PGx ha sido paralela a la medicina de precisión tanto en España como en el resto de los países. El desarrollo de la medicina de precisión en España ha estado muy influenciado por la existencia de planes y estrategias gubernamentales. Así en España se lanzó en 2020 la estrategia española de medicina personalizada2. Los gobiernos están realizando grandes inversiones, especialmente en infraestructuras como el programa IMPaCT43. Otro aspecto clave es la colaboración público-privada; en el área biomédica ésta es el resultado de los programas gubernamentales y del interés de las empresas privadas, que han visto el área como un buen negocio, así se entiende el crecimiento espectacular de empresas biotecnológicas en España (https://www.asebio.com/en). Uno de los resultados es el incremento de tratamientos biotecnológicos con la subida de los costos que supone y que se contrapone con la pretendida reducción tras la aplicación de la PGx. Para algunos44, esto puede generar desequilibrios sociales limitando el acceso al medicamento de las clases menos favorecidas.
La formación en PGx requiere especial atención ya que se necesita una masa crítica amplia para que la implementación se generalice. Como se ha explicado más arriba, algunos biomarcadores tienen una sólida evidencia científica y clínica, mientras que otros son solo asociaciones estadísticas, que se han demostrado falsas y con consecuencias clínicas. Para discriminar entre unos y otros es necesario contar con personas bien formadas. Tanto en farmacia como en medicina la formación en pregrado es muy escasa y en posgrado no hay una especialidad, sino cursos, algunos gratuitos, como los del consorcio COPHELA45. La tendencia es a formar equipos multidisciplinares en donde el farmacéutico debe jugar un papel importante dada su formación en farmacocinética y farmacodinamia. Las responsabilidades de los farmacéuticos en materia de PGx incluyen promover el uso óptimo y el momento oportuno de las pruebas PGx, interpretar los resultados de las pruebas PGx y educar a los profesionales sanitarios, los pacientes y el público en general sobre el campo de la farmacogenómica46,47. La creación de estos equipos en los hospitales es fácil y está mejorando con la creación de las unidades de medicina traslacional, pero puede ser un desafío en la farmacia comunitaria, no insalvable, gracias a las TIC.
El concepto de medicina de precisión ha ido evolucionando y con él, la PGx44. Inicialmente se hablaba de medicina personalizada, esto es, que cada persona recibiera el tratamiento más eficaz y evitando efectos secundarios. El concepto cambió al de medicina de precisión, en el que el objetivo ya no son solo los individuos sino también subgrupos de población y así evitar la posible mala interpretación del término personalizada. Asimismo, el concepto inicialmente basado en la genética ha evolucionado para incluir, además, la de otros factores. Las historias clínicas cada vez contienen más PGx junto con los llamados MAP (Medically Actionable Predispositions) o marcadores de predisposición médica23. Finalmente cabe destacar la tendencia hacia la medicina preventiva en donde la PGx se enfoca a la caracterización de poblaciones como anticipación a posibles tratamientos48. Un buen ejemplo es el proyecto GENOTRIAL del Hospital de la Princesa; la tendencia es general en todo el mundo49. Esta estrategia forma parte de otra más amplia a nivel mundial en la que se recogen todos los biomarcadores que muestran variación50. Esta nueva orientación ha sido acogida con enorme interés por la industria farmacéutica al seleccionar la población para los estudios clínicos.
ConclusionesLa aplicación de datos farmacogenéticos/farmacogenómicos en la clínica en España es comparable a otros países, y ha ido evolucionando conforme a la medicina de precisión. Los elementos clave para su desarrollo son la investigación traslacional, el apoyo gubernamental y la formación.
FinanciamientoNinguno.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
ContribucionesLos autores declaran que han contribuido por igual en el concepto, diseño, definición del contenido intelectual, búsqueda de la literatura, obtención y análisis de los datos y la preparación, edición y revisión del manuscrito.
Responsabilidad y cesión de derechosTodos los autores aceptamos la responsabilidad definida por el Comité Internacional de Editores de Revistas Médicas (disponible en http://www.icmje.org/).
Los autores cedemos, en el supuesto de publicación, de forma exclusiva los derechos de reproducción, distribución, traducción y comunicación pública (por cualquier medio o soporte sonoro, audiovisual o electrónico) de nuestro trabajo a Farmacia Hospitalaria y por extensión ala SEFH. Para ello se firmará una carta de cesión de derechos en el momento del envío del trabajo a través del sistema online de gestión de manuscritos.