la capecitabina es un fármaco antineoplásico utilizado en el tratamiento del cáncer de mama y de colon que puede dar lugar a una toxicidad grave, llegando a ser mortal en algunos pacientes. La variabilidad interindividual de esta toxicidad es debida en gran medida a las variaciones genéticas en los genes diana y las enzimas de metabolismo de este fármaco, como la timidilato sintasa y la dihidropirimidina deshidrogenasa. La enzima citidin desaminasa (CDA), imprescindible en la activación de la capecitabina, también presenta diversas variantes asociadas con un mayor riesgo de toxicidad al tratamiento, aunque su papel como biomarcador aún no está claramente definido.
Por ello, nuestro objetivo principal es estudiar la asociación entre la presencia de las variantes genéticas en el gen CDA, su actividad enzimática y el desarrollo de la toxicidad grave en los pacientes tratados con capecitabina, cuya dosis inicial se haya ajustado con base en el perfil genético del gen de la dihidropirimidina deshidrogenasa (DPYD).
Métodoestudio de cohortes observacional multicéntrico prospectivo, centrado en el análisis de la asociación genotipo-fenotipo de la enzima CDA.
Tras la fase experimental, se desarrollará un algoritmo que permita determinar el ajuste necesario de las dosis para disminuir el riesgo de toxicidad del tratamiento en función del genotipo CDA, elaborando una guía clínica para la dosificación de la capecitabina en función de las variantes genéticas en DPYD y CDA. Con base en esta guía, se creará una herramienta bioinformática que genere el informe farmacoterapéutico de manera automática, facilitando la implementación del consejo farmacogenético en la práctica clínica. Esta herramienta proporcionará un gran respaldo en la toma de decisiones farmacoterapéuticas basadas en el perfil genético del paciente, incorporando la medicina de precisión en la rutina clínica. Una vez validada la utilidad de esta herramienta, se ofrecerá de manera gratuita para facilitar la implementación de la farmacogenética en los centros hospitalarios y beneficiar de forma equitativa a todos los pacientes en tratamiento con capecitabina.
Capecitabine, an antineoplastic drug used in the treatment of breast and colon cancer, can cause severe, even fatal toxicity in some patients. The interindividual variability of this toxicity is largely due to genetic variations in target genes and enzymes of metabolism of this drug, such as thymidylate synthase and dihydropyrimidine dehydrogenase. The enzyme cytidine deaminase (CDA), involved in the activation of capecitabine, also has several variants associated with an increased risk of toxicity to treatment, although its role as a biomarker is not yet clearly defined.
Therefore, our main objective is to study the association between the presence of genetic variants in CDA gen, CDA enzymatic activity and the development of severe toxicity in patients treated with capecitabine whose initial dose was adjusted based on the genetic profile of the dihydropyrimidine dehydrogenase gen (DPYD).
MethodProspective multicenter observational cohort study, focused on the analysis of the genotype-phenotype association of the CDA enzyme.
After the experimental phase, an algorithm will be developed to determine the dose adjustment needed to reduce the risk of treatment toxicity according to CDA genotype, developing a clinical guide for capecitabine dosing according to genetic variants in DPYD and CDA. Based on this guide, a Bioinformatics Tool will be created to generate the pharmacotherapeutic report automatically, facilitating the implementation of pharmacogenetic advice in clinical practice. This tool will be a great support in making pharmacotherapeutic decisions based on the patient's genetic profile, incorporating precision medicine into clinical routine. Once the usefulness of this tool has been validated, it will be offered free of charge to facilitate the implementation of pharmacogenetics in hospital centers and equitably benefit all patients on capecitabine treatment.
La capecitabina, del grupo de las fluoropirimidinas, es un fármaco antineoplásico utilizado en el tratamiento del cáncer de mama y de colon, que da lugar a una toxicidad grave en un 25,5% de los pacientes tratados1, llegando a producir la muerte en un 1,6% de los mismos2. Una de las causas de la variabilidad interindividual de los efectos tóxicos de este fármaco son las variantes genéticas en las enzimas implicadas en su metabolismo, así como en sus genes diana.
El gen que codifica la enzima timidilato sintasa es un potencial marcador de respuesta y de toxicidad a las fluoropirimidinas, ya que el mecanismo de acción principal de estos fármacos es la inhibición del ciclo del folato a través del bloqueo de esta enzima. Se han estudiado diversas variantes en este gen, observándose una asociación significativa entre el genotipo 2R/2R de la variante rs45445694 y la toxicidad grave al tratamiento (p=0,0014; OR=5,21). Además, el análisis de la expresión en los tejidos tumorales sugiere una correlación entre este genotipo y la baja expresión de timidilato sintasa3.
La dihidropirimidina deshidrogenasa (DPD) es la enzima limitante del catabolismo de las fluoropirimidinas. Existe una amplia evidencia de que la deficiencia de DPD está asociada con un aumento de los niveles del metabolito activo en la sangre, provocando una toxicidad severa tras la administración del fármaco que puede llegar a ser mortal4. Las variantes con función disminuida de mayor relevancia en el gen que codifica la DPD (DPYD) son 4: DPYD*2A o c.1905+1G>A (rs3918290); DPYD*13 o c.1679T>G (rs55886062); c.2846A>T (rs67376798) y c.1129-5923C>G o HapB3 (rs75017182)5,6. La recomendación por parte de la Agencia Española del Medicamento y Productos Sanitarios (AEMPS) de ajuste de las dosis de fluoropirimidinas se apoya en la Guía del Consorcio de Implementación de Farmacogenética Clínica7, en la Guía del Grupo de Trabajo Holandés de Farmacogenética8 y en el consenso de expertos de la Sociedad Española de Farmacogenética y Farmacogenómica y de la Sociedad Española de Oncología Médica9. En estas guías, se establece un estado metabólico para cada genotipo con la correspondiente recomendación respaldada en una evidencia de nivel uno de alta calidad.
Diversas variantes en el gen que codifica la enzima citidin desaminasa (CDA), imprescindible en la activación del fármaco, también se han asociado con un aumento en la toxicidad de la capecitabina. Concretamente, las variantes 79 A>C (rs2072671)10–12; 451 A>G (rs532545)10,13–15; -33 ins/delC13; 92 A>G (rs602950)15 y 435 C>T (rs1048977)11 de CDA se han asociado con un mayor riesgo de toxicidad al tratamiento, observándose en algunas de ellas una alteración de la actividad enzimática16.
Sin embargo, a pesar de todas las evidencias publicadas, no existe una guía clínica para la dosificación de este fármaco en función de las variantes genéticas en CDA, por lo que es necesario establecer el valor pronóstico real de este biomarcador mediante los estudios de la asociación genotipo-fenotipo. Además, cabe destacar que casi un 60% de la población europea es portadora de alguna de estas variantes genéticas11,16, por lo que un elevado porcentaje de la población se vería beneficiado de un cribado genético de este biomarcador previo al inicio del tratamiento con capecitabina.
En cuanto a la justificación económica, el gasto medio de las consultas, hospitalizaciones y tratamientos derivados de los efectos adversos por la capecitabina es aproximadamente de 500 € por paciente según un estudio realizado en el Reino Unido17. Estos datos son extrapolables a la población española, puesto que se trata de la misma etnia y del mismo tipo de modelo sanitario. Además, se estima que el cribado del genotipo de DPYD y CDA en la rutina clínica podría reducir en un 50% estos costes18. Teniendo en cuenta el número de los pacientes tratados con capecitabina en los centros participantes, aproximadamente 80 pacientes al año, la implementación del genotipado de DPYD y CDA supondría un ahorro de unos 20.000 € al año por centro. Si extrapolamos estos datos al conjunto de la población española, este ahorro sería de 5.000.000 € al año.
El impacto que supondría incluir CDA en la guía de dosificación radica en la alta frecuencia de mutación de este gen. Según la base de datos dbSNP del National Center for Biotechnology Information, las 4 variantes más relevantes en DPYD presentan una frecuencia alélica media de 0,005 en la población europea, mientras que la frecuencia media de las 5 variantes seleccionadas en CDA es de 0,355, por lo que el ahorro correspondiente al cribado de CDA podría implicar un 98,6% (4.930.000 €) del ahorro anual estimado, aumentando notablemente la sostenibilidad del Sistema Nacional de Salud si se compara con el cribado único de DPYD que se realiza actualmente.
El objetivo de este estudio es analizar la asociación entre la presencia de las variantes genéticas en la enzima CDA, la actividad enzimática de la misma y el desarrollo de toxicidad grave (grado 3-4 de diarrea, nauseas/vómitos, mucositis, síndrome de eritrodisestesia palmoplantar, hepatotoxicidad y/o toxicidad hemática) en los pacientes que inicien el tratamiento con capecitabina.
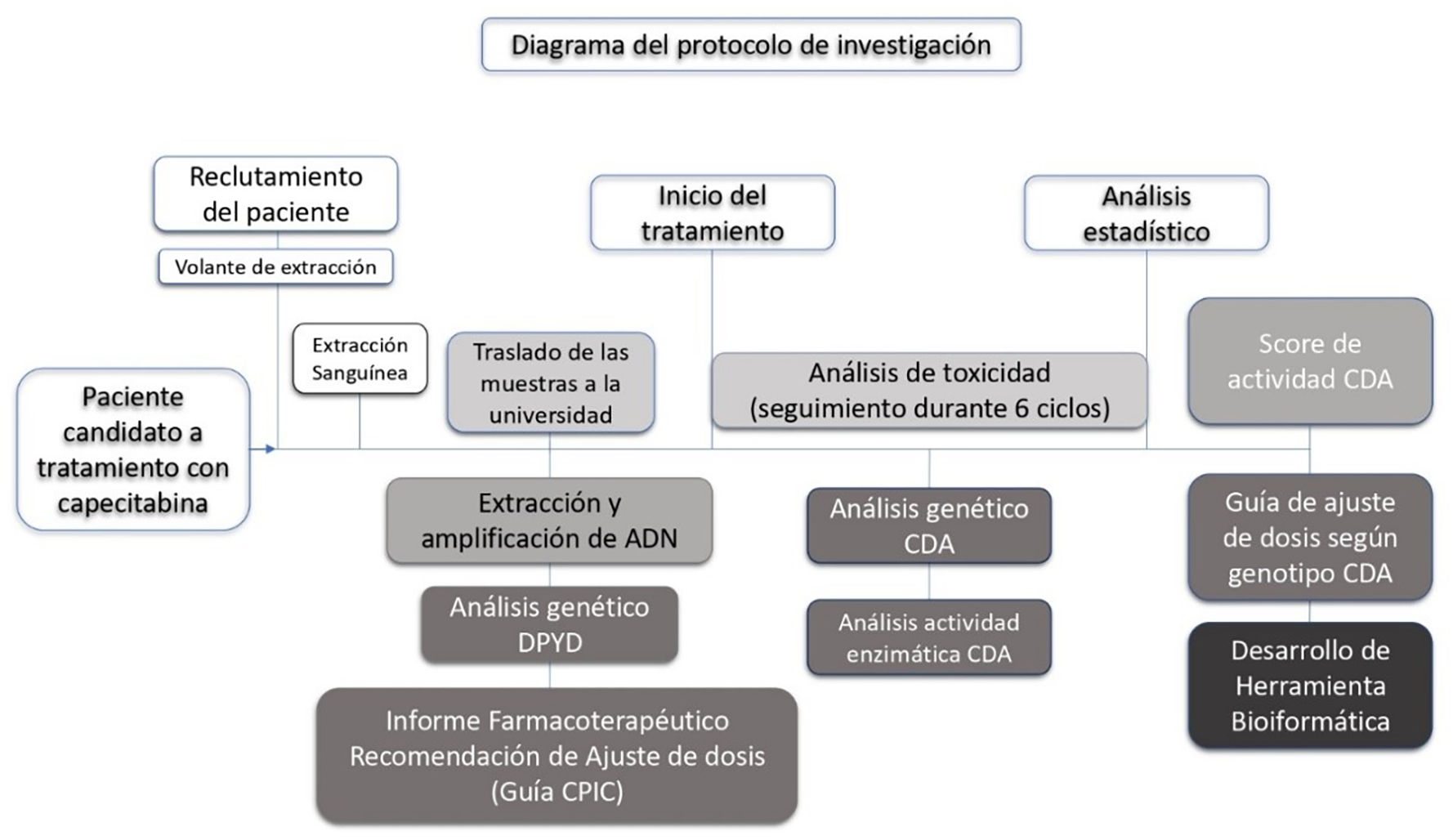
Además, en el estudio se incluye el análisis de las 4 variantes genéticas en DPYD recomendadas por la AEMPS: DPYD*2A o c.1905+1G>A (rs3918290); DPYD*13 o c.1679T>G (rs55886062); c.2846A>T (rs67376798) y c.1129–5923C>G o HapB3 (rs75017182)19. Este genotipado se realizará previamente al tratamiento con capecitabina, permitiendo el ajuste de la dosis inicial en función del genotipo DPYD del paciente. Posteriormente, se realizará el estudio de las frecuencias alélicas de las variantes analizadas, el estudio de la frecuencia y el grado de toxicidad de los participantes, y la determinación del ajuste necesario de la dosis en función de las variantes genéticas en CDA para la reducción de la toxicidad al tratamiento. Tras la validación de los resultados obtenidos en una cohorte independiente, se elaborará una guía clínica de dosificación de capecitabina en función de las variantes genéticas en DPYD y CDA y se desarrollará una herramienta bioinformática que asocie el genotipo de DPYD y CDA del paciente con el ajuste de las dosis recomendado en la guía clínica de dosificación. En la figura 1 se puede observar el diagrama de flujo con las diferentes etapas del protocolo de investigación.
MétodoDiseñoEstudio observacional prospectivo multicéntrico analítico de cohortes, en el cual la variable de exposición es la presencia/no presencia de las variantes genéticas en CDA y las variables respuesta son la toxicidad grave/leve y la actividad enzimática de CDA.
Ámbito de la aplicaciónLa población del estudio incluye pacientes que inicien el tratamiento con capecitabina del Hospital General Universitario de Elda, del Hospital General Universitario de Elche y del Hospital Virgen de los Lirios de Alcoy, durante un periodo de selección de 2 años.
Duración del estudioLa duración total estimada para la realización del estudio es de 4 años: 2 años para el periodo de selección, un año para la validación de los resultados en cohortes independientes y un año para el desarrollo de la Guía Clínica de Dosificación y la herramienta informática.
Criterios de selecciónCriterios de inclusión: pacientes mayores de 18 años que inicien el tratamiento con capecitabina y acepten participar en el estudio mediante el consentimiento informado.
Criterios de exclusión: pacientes que no acepten participar en el estudio.
IntervencionesLa intervención se basará en el análisis de las variantes genéticas en DPYD y CDA y el análisis de la actividad de la enzima CDA a partir de extracción sanguínea rutinaria antes del inicio del tratamiento. El tratamiento farmacológico será el establecido en la práctica clínica habitual, contando con el informe de los polimorfismos en DPYD para realizar el ajuste de las dosis previo al inicio del tratamiento, según las guías clínicas publicadas, en los pacientes portadores de alguna de las variantes de riesgo.
Tamaño de la muestraPuesto que no se conoce el riesgo relativo (RR) de desarrollar la toxicidad grave en los portadores de estas variantes, ya que los estudios publicados hasta la fecha no son de cohortes sino de casos y controles, se realizará en primer lugar una muestra piloto, analizando las variables principales en 30 pacientes expuestos y 30 pacientes no expuestos. En segundo lugar, se calculará el tamaño muestral necesario para realizar el estudio teniendo en cuenta el RR obtenido en la muestra piloto y el riesgo de desarrollar el desenlace en el grupo no expuesto, asumiendo, según la evidencia científica publicada, una hipótesis unilateral con un error α o tipo I del 5% y un error β ο tipo II del 20% (nivel de confianza del 95% y potencia del 80%).
Teniendo en cuenta que la media de selección anual es de 80 pacientes por centro, tratándose de un estudio multicéntrico con 3 centros participantes, se podrían seleccionar aproximadamente 240 pacientes al año, por lo que se estima suficiente un periodo de selección de 2 años. Si tras el análisis de la muestra piloto se determina necesario un tamaño muestral superior al obtenido en el periodo de selección, se propondrá la inclusión de nuevos centros participantes en el estudio, aumentando considerablemente la velocidad de la selección.
VariablesLas variables principales son 2: en primer lugar, la incidencia de toxicidad grave en el grupo expuesto y el grupo no expuesto. Se considera toxicidad grave un grado 3 o superior de alguno de los siguientes efectos adversos clasificados según los criterios CTCAE 5.0: diarrea, nauseas/vómitos, mucositis, síndrome de eritrodisestesia palmoplantar, hepatotoxicidad, toxicidad hematológica y toxicidad global, pudiendo registrarse otros tipos de toxicidad no contemplados entre los anteriores. Se trata de una variable cualitativa de categorización binaria.
En segundo lugar, la actividad enzimática de CDA en los grupos expuesto y no expuesto, que se determinará tras la validación del método espectrofotométrico descrito previamente por otros autores16. Se trata de una variable cuantitativa continua, expresada en unidad arbitraria (AU)/mg de proteína, que representa μmol de amoníaco/h/mg de proteína.
La variable de exposición es la presencia de variantes genéticas en CDA. Se trata de una variable cualitativa de categorización binaria: portador (paciente homocigoto mutante o heterocigoto)/no portador (paciente homocigoto wild type). El grupo expuesto estará formado por los pacientes portadores de alguna de las siguientes variantes genéticas en CDA: 79 A>C (rs2072671); 451 A>G (rs532545); -33 ins/delC (rs3215400), 92 A>G (rs602950) y 435 C>T (rs1048977).
Grupos del estudioEl grupo de pacientes expuestos se formará por los pacientes portadores de alguna de las variantes anteriores, mientras que el grupo de pacientes no expuestos incluirá a los pacientes no portadores de ninguna de las variantes.
Recogida de datosLa selección de los pacientes se realizará por parte del Servicio de Oncología, incluyendo a todos los pacientes que inicien el tratamiento con capecitabina en los centros participantes y que acepten participar en el estudio. En el momento de la inclusión, tras la entrega de la hoja de información al paciente y la firma del consentimiento informado, se cumplimentará la entrevista inicial y se programará la extracción sanguínea previa al inicio del tratamiento.
El oncólogo/a prescriptor/a avisará, mediante el volante de petición, al Servicio de Análisis Clínicos del hospital, indicando la fecha de extracción y el número de la historia clínica del paciente. Tras el análisis bioquímico, el tubo de sangre total en EDTA (2 ml) y el tubo de suero (2 ml) serán almacenados a 4 y -20 °C respectivamente en el laboratorio central hasta su recogida para proceder al análisis genético y enzimático.
La recogida de las muestras se efectuará semanalmente para su traslado al Laboratorio de Toxicología y Medicina Legal ubicado en el campus de San Juan de la Universidad Miguel Hernández de Elche. Allí se realizará la extracción y la purificación de ADN y el análisis del genotipo de las 4 variantes en DPYD recomendadas por la AEMPS, mediante PCR en tiempo real con sondas TaqMan en termociclador Step One™, realizándose el análisis de resultados con software StepOne v2.3.
El informe farmacoterapéutico con la recomendación del ajuste de dosis se remitirá al día siguiente mediante correo electrónico al servicio de farmacia de los centros participantes. Este servicio incluirá el informe en la historia clínica del paciente a través del programa informático del centro, permitiendo que el oncólogo/a correspondiente tenga acceso al mismo y se pueda iniciar el tratamiento con la pauta adecuada.
Una vez iniciado el tratamiento, las muestras de ADN se congelarán a -20 °C hasta la realización del análisis de las 5 variantes genéticas en CDA mediante PCR en tiempo real.
Las muestras de suero serán almacenadas a −80 °C hasta que se realice el análisis de la actividad enzimática por espectrofotometría.
Los datos clínicos de toxicidad al tratamiento serán recogidos en la Hoja de Recogida de Datos Electrónica por parte del oncólogo/a prescriptor/a durante los 6 primeros ciclos del tratamiento, siguiendo los criterios CTCAE 5.0.
Una vez alcanzado el tamaño muestral necesario con base en la muestra piloto, se procederá al análisis estadístico. Con los resultados obtenidos se realizará un estudio de asociación entre los datos genéticos y enzimáticos, desarrollando un algoritmo que determine el estado metabólico para cada diplotipo posible, con la combinación de las 5 variantes en CDA y el riesgo de toxicidad asociado.
Tras la validación de estos resultados en una cohorte independiente, se obtendrá una evidencia científica de peso que determine el ajuste necesario de dosis para reducir el riesgo de toxicidad en cada genotipo concreto, permitiendo la elaboración de una guía clínica con base en el genotipo en DPYD y CDA.
Para facilitar la implementación del genotipado en los hospitales españoles, se desarrollará una herramienta bioinformática mediante el software AlleleTyper™ donde se aplicará el algoritmo. Esta herramienta se basará en la asociación del diplotipo del paciente con el estado metabólico y la recomendación de ajuste de dosis, permitiendo la generación automática del informe farmacoterapéutico. Una vez validada la utilidad de esta herramienta en cohortes independientes, se ofrecerá de manera gratuita facilitando su implementación en los centros hospitalarios para poder beneficiar de forma equitativa a todos los pacientes en tratamiento con capecitabina.
Análisis estadísticoSe realizará un análisis descriptivo bivariante y multivariante. El análisis descriptivo se realizará según el tipo de variante. De las variantes principales, es decir, la incidencia de toxicidad grave y la actividad enzimática de CDA, se calcularán los límites de confianza al 95%.
En el análisis bivariante, se comprobará la distribución de la normalidad y si es comprobada, se utilizarán las pruebas paramétricas; en caso contrario, se utilizarán las pruebas no paramétricas. Según los tipos de variables, se utilizarán distintas pruebas paramétricas: Chi-cuadrado para comparar 2 variables cualitativas, t de Student para comparar una variable cualitativa y una variable cuantitativa, y la correlación lineal Pearson para comparar 2 variables cuantitativas. La significación estadística se fija en p=0,05.
En cuanto al análisis multivariante, como la variable dependiente es cualitativa, se utilizará la regresión logística binaria y por pasos, calculando el RR con límite de confianza del 95%. Los análisis se realizarán mediante el programa SPSS v.26 y el programa R v.4.0.2 por parte del Servicio de Estudios Estadísticos de Fisabio, en España.
DiscusiónLa innovación de este proyecto de asociación genotipo-fenotipo, mediante la categorización de la enzima CDA como biomarcador, consiste en la recomendación de la prescripción farmacológica personalizada con base en el genotipo DPYD y CDA de cada paciente, y no solo en DPYD, como se recomienda actualmente.
Tras la validación en una cohorte independiente de la guía clínica que se pretende desarrollar, se espera reducir el riesgo de toxicidad del tratamiento con capecitabina debido a la sobredosificación en el caso de los pacientes portadores de las variantes genéticas en CDA de metabolismo ultrarrápido y/o los portadores de las variantes genéticas en DPYD de metabolismo pobre.
Una de las principales diferencias de este proyecto con respecto a los ya publicados es su diseño prospectivo de cohortes. La gran mayoría de los estudios son retrospectivos, presentan mayores limitaciones y observan toxicidades para asociar el polimorfismo con la toxicidad, sin realizar un ajuste de dosis11,20,21. En el estudio actual, las determinaciones y el consejo farmacogenético se realizan de forma prospectiva, aprovechando los recursos y el beneficio clínico de la realización del genotipado preventivo. Además, el análisis estadístico nos proporcionará el RR de la presencia de las variantes en CDA, siendo esta una medida estadística mucho más potente que las obtenidas en estudios de casos y controles. También, cabe tener en cuenta que, gracias al desarrollo de la herramienta bioinformática, se facilitará la implementación del Servicio de Análisis Farmacogenético e interpretación de los resultados en la práctica clínica de los centros hospitalarios.
Finalmente, la categorización de CDA como biomarcador podrá ser útil también en el tratamiento con otros fármacos, ya que esta enzima está implicada en la inactivación de la gemcitabina y la citarabina, antineoplásicos utilizados para diversos tipos de cáncer. Así pues, los resultados obtenidos en este proyecto se podrán utilizar en posteriores estudios con el objetivo de elaborar guías clínicas de dosificación para dichos fármacos y reducir la quimiorresistencia al tratamiento y la consecuente progresión de la enfermedad.
LimitacionesEl hecho de analizar únicamente la actividad enzimática para establecer el fenotipo metabólico puede suponer una limitación, puesto que también es importante conocer la farmacocinética de cada paciente para describir un fenotipo más completo frente al metabolismo de la capecitabina. Esta sería una futura línea de investigación, ampliar la caracterización de CDA como biomarcador incluyendo al fenotipado el análisis farmacocinético durante las primeras dosis de capecitabina de cada paciente. En este sentido, se ha estudiado recientemente a través de un modelo farmacocinético poblacional, la relación entre los metabolitos de la capecitabina (5-FU, 5'-DFCR y 5'-DFUR) y la toxicidad o la respuesta clínica al fármaco22. Los datos obtenidos relacionan a la enzima CDA como covariable estadísticamente significativa, lo cual pone de manifiesto la conveniencia de analizar simultáneamente los datos genéticos, enzimáticos y farmacocinéticos de este biomarcador.
Responsabilidades éticasEste proyecto ha sido clasificado por la Agencia Española del Medicamento y Productos Sanitarios, con código ATB-CAP-2020-01, como «Estudio Postautorización de seguimiento prospectivo (EPA-SP)» y ha obtenido la autorización por parte del Comité Autonómico de Evaluación de Estudios Postautorización Observacionales, de Seguimiento Prospectivo con medicamentos (CAEPO) así como el dictamen favorable del Comité de Ética de Investigación con Medicamentos (CEIM) del Hospital General Universitario de Elda y del Hospital General Universitario de Elche de acuerdo a la legislación vigente, el Real Decreto 223/2004 del 6 de febrero.
Financiación- -
V Convocatoria de Ayudas para el Fomento de la Actividad Investigadora de la Fundación para el Fomento de la Investigación Sanitaria y Biomédica de la Comunidad Valenciana (FISABIO) 2021.
- -
Ayudas para Acciones Preparatorias de apoyo a la exploración y formulación de futuros proyectos de investigación y de innovación conjuntos entre Universidad Miguel Hernández de Elche y FISABIO 2021.
- -
VIII Convocatoria de ayudas para el Apoyo y Fomento de la Investigación del Instituto de Investigación Sanitaria y Biomédica de Alicante (ISABIAL) 2021, modalidad B.
- -
Patrocinio Roche con código CPRES00254.
La contribución de cada uno de los autores en el trabajo y en la elaboración del artículo es la siguiente:
Paula Castro-Sánchez: promoción del estudio, elaboración del protocolo, solicitud de clasificación y autorizaciones necesarias para la aprobación del estudio en los centros participantes, coordinación general del estudio, redacción del artículo.
Andrés Corno-Caparrós: diseño, organización y ejecución del análisis genético del estudio, planteamiento y desarrollo de herramienta bioinformática, revisión crítica del artículo y aprobación de la versión final para su publicación.
M. Amparo Talens-Bolós: diseño y organización de la logística de muestras entre los centros hospitalarios participantes y la universidad, emisión de informes farmacogenéticos, revisión crítica del artículo y aprobación de la versión final para su publicación.
María José Prieto-Castelló: solicitud de ayudas públicas para la financiación del estudio, gestión de datos para el análisis estadístico, revisión crítica del artículo y aprobación de la versión final para su publicación.
Loreto Pitaluga-Poveda: validación del método de análisis de la actividad enzimática, gestión y preparación de reactivos, revisión crítica del artículo y aprobación de la versión final para su publicación.
Juan Antonio Barrera-Ramírez: organización y realización del reclutamiento de pacientes y la recogida de datos clínicos, revisión crítica del artículo y aprobación de la versión final para su publicación.
Conflicto de interesesNo existe ningún conflicto de intereses.
AgradecimientosA todos los profesionales de los servicios de farmacia hospitalaria, oncología médica y análisis clínicos de los centros participantes que colaboran en el desarrollo de este proyecto. Al Laboratorio de Análisis Genéticos ANCOR por su apoyo a nivel técnico e intelectual que ha permitido el desarrollo de este estudio.